
Rで非線形回帰分析&ggplot2で描画:対数近似の例(R)
- Part 1:データの整形(tidyr::gathar())
- Part 2:非線形回帰分析(nls()&glm()で対数近似・パラメータ推定)
- Part 3:ggplot2で描画(複数のデータセットを重ね描き)
Part 3:ggplot2で描画(複数のデータセットを重ね描き)
○ まずは平均値と95%信頼区間をstat_smooth()でプロット
ggplot2ではstat_smooth()やgeom_line(stat="smooth")で近似曲線を描くことができます。
Part2では、glm()やnls()でパラメータ推定を行い、PSEやJNDの算出を行いました。
ここでは、ggplot2で描いた近似曲線上へ、glm()などで算出したPSEやJNDの点をプロットします。
算出に使った式を、ggplot2の描画関数にも適用する必要があります。
まずはggplot2のstat_smooth()とPart 1で作った平均値のデータフレームを用いて、群ごとに各条件の平均値へ近似曲線をフィッティングします。
その際、geom_point()で各条件でのデータをプロットします。
g1 <- ggplot(data = d2, aes(x = duration, y = rate, color = group, fill = group))+
geom_point(size = 3)+
stat_smooth(method = "glm", formula =y ~ log(x), se = TRUE, size = 2)
plot(g1)
ggsave("fitdemo1.png",g1)
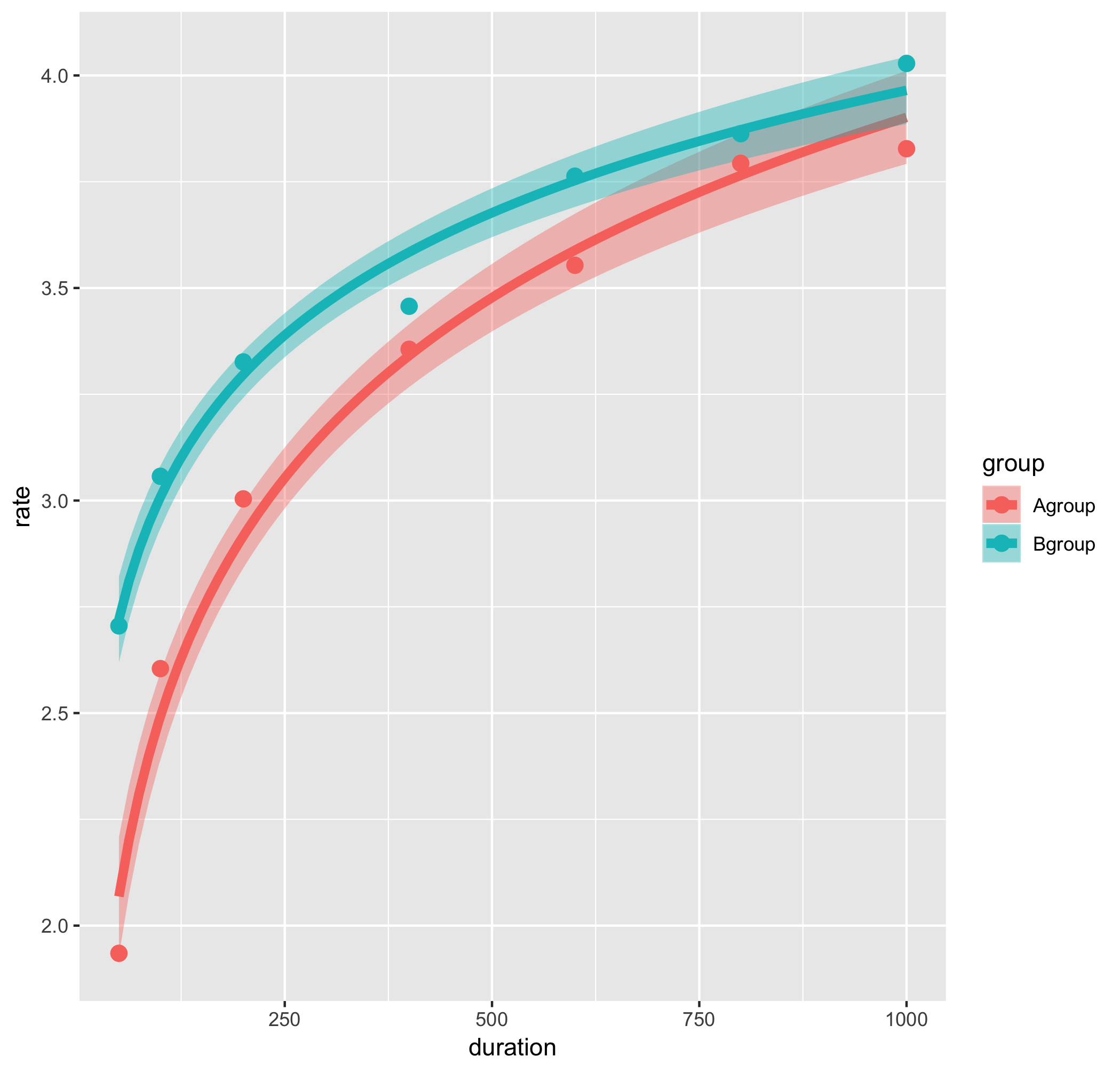
ggplot()で描画するggplotオブジェクトを初期化します。使うデータフレームや、aes()でプロットするデータ、色分けのキーとなる列(ここではgroup)をここで指定すると、その後「+」で継ぎ足されていく他の関数でもそれらの設定が使用されます。
geom_point()でスカッタープロット、その上にstat_smooth()で近似曲線と信頼区間を描いています。
se=FALSEとすると信頼区間の帯が非表示に、alpha=を0~1で指定すると帯の透明度を変更できます。
さて、Part 2で推定したパラメータを用いて、それぞの近似曲線上にJNDを打ってみたいと思います。
現段階で一番簡単な方法は、annotate()でテキストとして任意の座標上へ点を打つことです。
ggplot2では、すでにワークスペース上にあるggplotオブジェクトへ、以下のように「+」で新たな情報を継ぎ足すことができます。
A <- d2[d2$group=="Agroup",]
B <- d2[d2$group=="Bgroup",]
fitA <- glm(rate ~ log(duration), family=gaussian, data=A)
fitB <- glm(rate ~ log(duration), family=gaussian, data=B)
slopeA <- summary(fitA)$coefficients[2]
interceptA <- summary(fitA)$coefficients[1]
slopeB <- summary(fitB)$coefficients[2]
interceptB <- summary(fitB)$coefficients[1]
jnd <- max(d$rate) * 0.75 #便宜上、全ローデータ中の75%点をJNDとしています。
sA <- exp(1)^((jnd - interceptA) / slopeA)
sB <- exp(1)^((jnd - interceptB) / slopeB)
g1 <- g1 + annotate("text", x = sA, y = jnd, label = "+", size = 10)+
annotate("text", x = sB, y = jnd, label = "+", size = 10)
plot(g1)
ggsave("fitdemo2.png",g1)
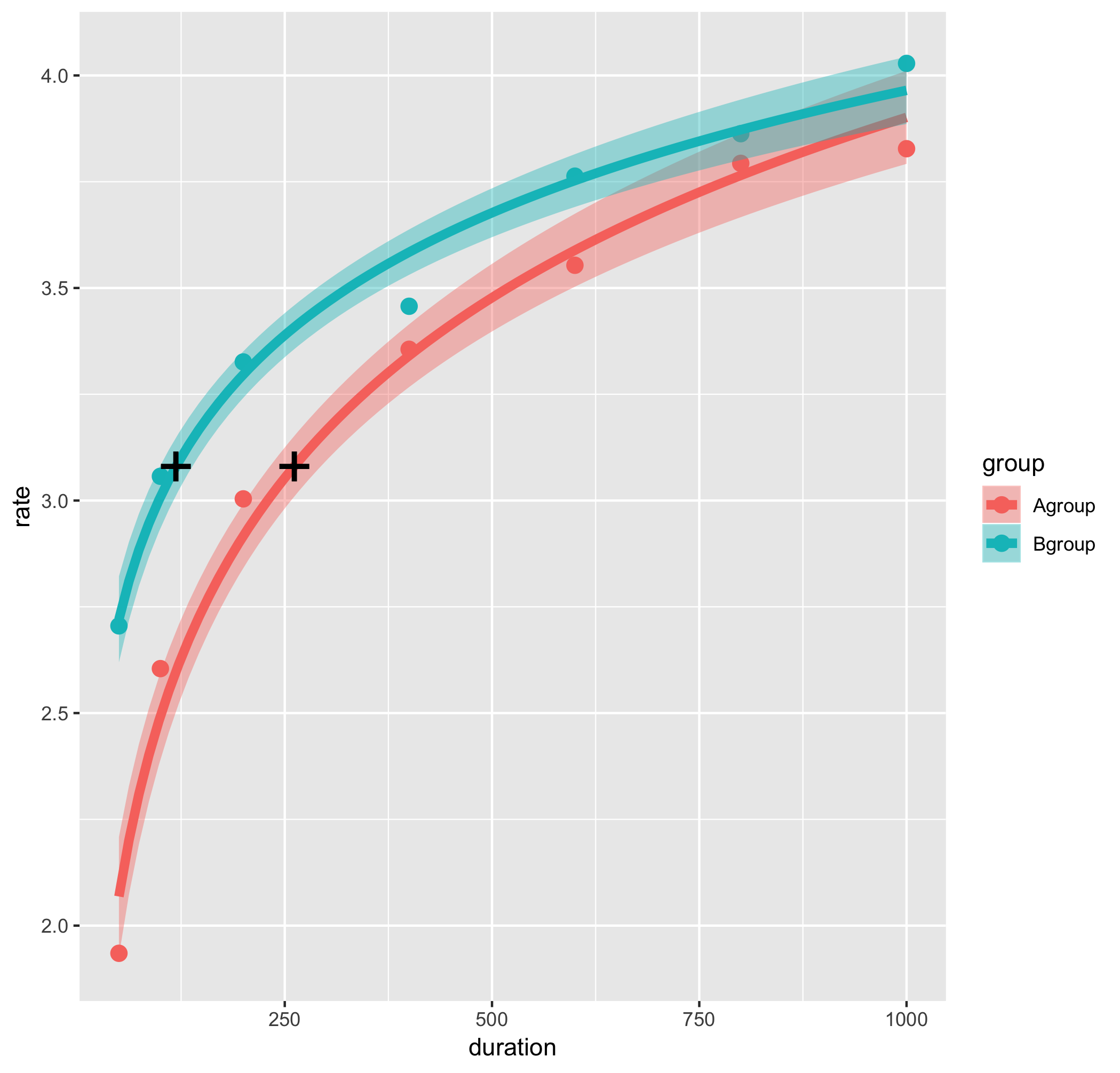
うまく近似曲線上にプロットできたようです。
ですが、所詮これはテキストで描画しているので、見た目の統一感に欠けます。
別の方法は、カーブフィッティング用のデータと分けてしまうことです。
さきほどggplot()で指定したデータセットに従ってプロットが描出されてしまうため、ggplotオブジェクトを別途作成する必要があります。
まずは、各群のJNDを収めたデータフレームを作成します。
m <- data.frame(group = c("Agroup", "Bgroup"), JND = c(sA,sB), coln = c("col1", "col2"))
そうしたら、今度はggplot()以降の関数でそれぞれデータを指定しオブジェクトを生成します。
g1 <- ggplot()+
stat_smooth(data = d2, aes(x = duration, y = rate, color = group, fill = group), method = "glm", formula =y ~ log(x), se = TRUE, size = 2)+
geom_point(data = d2, aes(x = duration, y = rate, color = group), size = 3)+
geom_point(data = m, aes(x = JND, y = jnd, color = group), size = 10, shape = 3)
plot(g1)
ggsave("fitdemo3.png",g1)
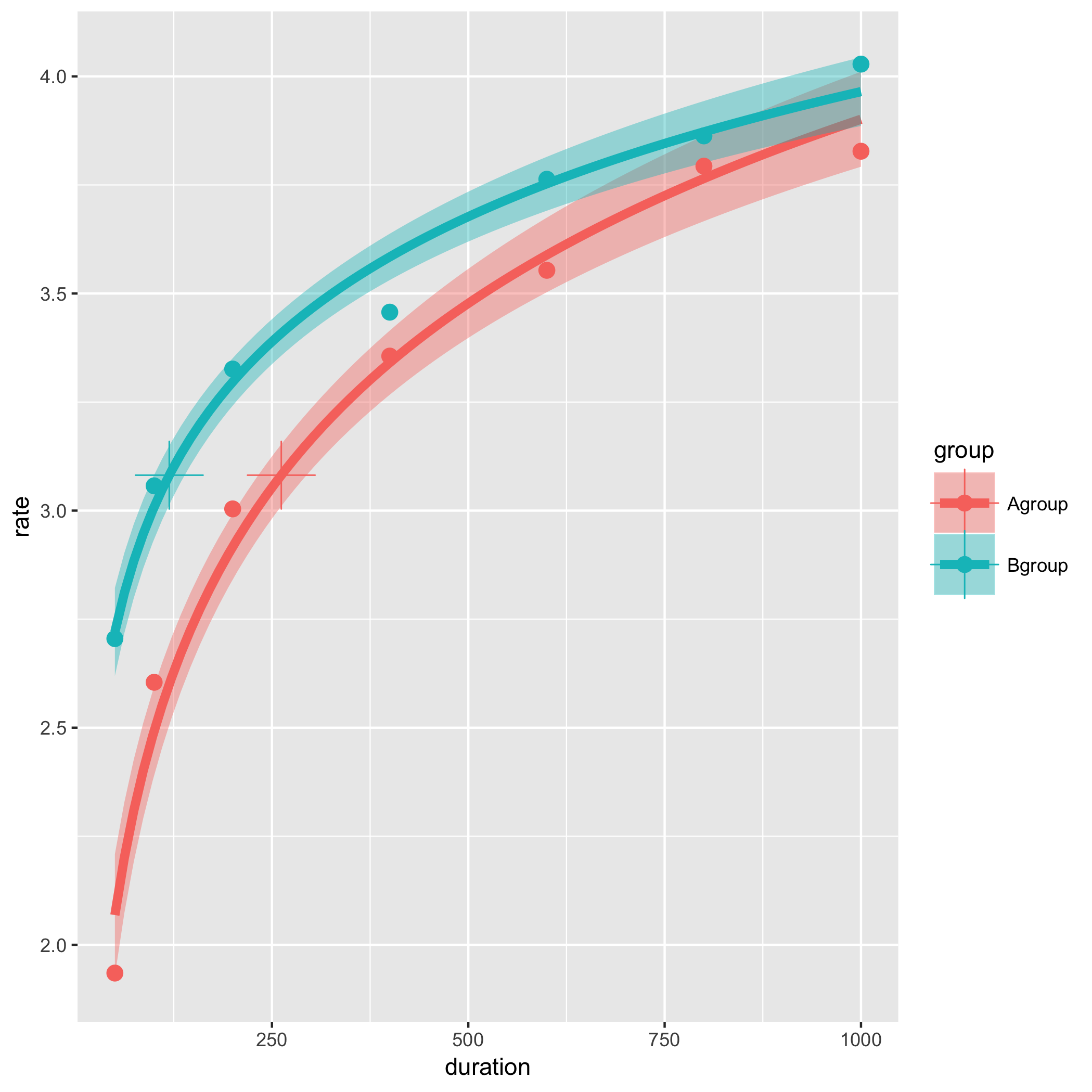
geom_point()の引数shapeでパッチ(pch)の種類を変えてみました。
上の例では、データポイントは各条件の平均値1つずつから算出しています(なのでパッチ「+」はエラーを表しているわけではありません)。
ですが、被験者間での誤差や95%信頼区間を算出したいこともあると思います。
さらに、群ごとの信頼区間ではなく、各被験者のフィッティングカーブを描出したい場合もあるでしょう。
○ 被験者ごとに近似曲線を描く
PSEs <- NULL
JNDs <- NULL
pse <- max(d$rate)*0.5
jnd <- max(d$rate)*0.75 #便宜上、全ローデータ中の75%点をJNDとしています。
subids <- names(table(d$ID)) #被験者ID
g1 <- ggplot() #ggplotオブジェクトを初期化
for(i in 1:length(subids)){
subd <- d[d$ID == subids[i], ] #被験者IDごとのデータセットを作成
g1 <- g1 + stat_smooth(data = subd, aes(duration, rate, color = group), method = "glm", formula = y ~ log(x), se = FALSE)+
geom_point(data = subd, aes(duration, rate, color = group)) #各被験者の近似曲線とスカッタープロットを継ぎ足していく。
fit <- glm(rate ~ log(duration),family = gaussian, data = subd)
a <- as.numeric(fit$coefficients[2])
b <- as.numeric(fit$coefficients[1])
dp <- exp(1)^((pse - b) / a)
s <- exp(1)^((jnd - b) / a)
PSEs <- c(PSEs, dp)
JNDs <- c(JNDs, s)
}
plot(g1)
ggsave("fitdemo4.png", g1)
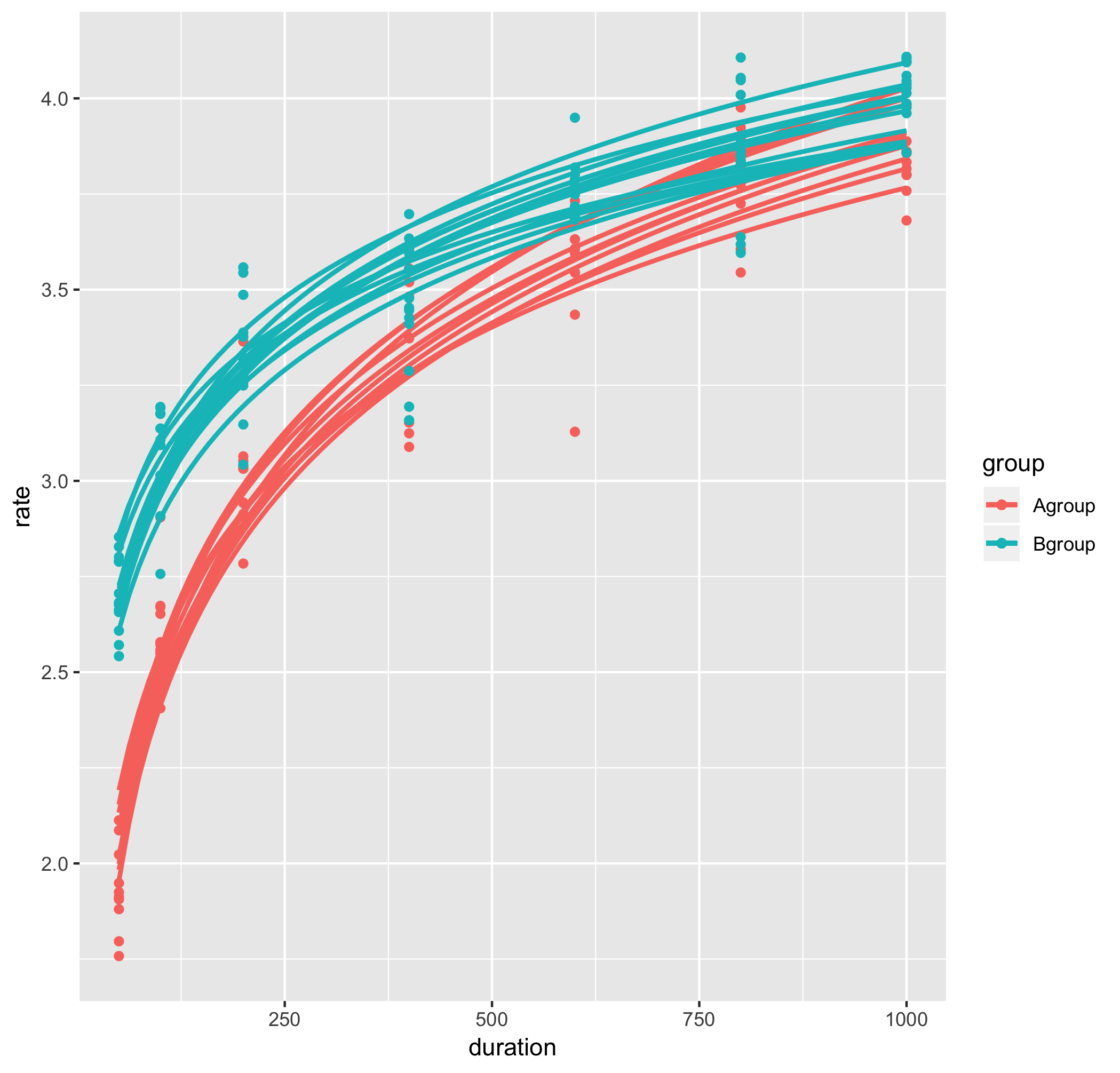
これで、変数JNDsとPSEsに被験者IDの順番でそれぞれのデータが格納されました。
○ 異なるデータセットでグラフを重ね描きする
平均値と、ついでに95%信頼区間を算出、新たなデータフレームに収めます。
ci <- function(y) qt(0.975,length(y)-1)*sd(y)/sqrt(length(y)) # 信頼区間を計算する関数を定義
d3 <- data.frame(
group = c("Agroup", "Bgroup"),
meanP = c(mean(PSEs[1:10]), mean(PSEs[11:23])),
meanJ = c(mean(JNDs[1:10]), mean(JNDs[11:23])),
ciP = c(ci(PSEs[1:10]), ci(PSEs[11:23])),
ciJ = c(ci(JNDs[1:10]), ci(JNDs[11:23])),
coln = c("col1", "col2")
)
これで、今度は群ごとの平均値とCIが収められたデータフレームができました。
群ごとの各条件での反応率が収められたd2、算出した平均値と信頼区間が格納されたd3を追加して、重ね描きしたいと思います。
ところで、このままでは平均値と個々人のプロットとで、カラーコードがかぶってしまいます。
そこで、文字列と色名を新たに対応させ、区別することにします。
cols <- c("Agroup" = "#F8766D", "Bgroup" = "#00BFC4", #ggplot2のデフォルト色
"col1" = "indianred4", "col2" = "darkcyan"
)
これで、グループ名を色のキーに指定した場合、各グループ名(文字列)に対応した色が呼び出され、データフレームを作成した際に追加した色名の列を指定すれば、それに対応した色が呼び出されます。
下記のサイトを参考に、ggplot2のデフォルトカラーを設定しました。
ggplot2のデフォルトの色を知りたい
さてここで、先程作ったggplotオブジェクトに平均値のプロットを継ぎ足したいと思います。
したがって、群ごとに各条件の平均値を収めたd2と、群ごとに被験者間のJNDの平均値とCIを収めたd3を用います。
せっかくJNDの信頼区間を計算したので、エラーバーをつけます。
geom_errorbar()を使うとy軸方向(垂直)にエラー範囲をつけられますが、x軸方向(水平)にはgeom_errorbarh()を使います。
先程指定した色名を適用するためには、手動で色を設定する関数scale_color_manual()を使います。この引数valuesに色名を収めたベクトルを指定します。
信頼区間の帯の色は、scale_fill_manual()で指定します。
g1<-g1+stat_smooth(data=d2,aes(duration,rate,color=coln),method="glm",formula=y~log(x),se=FALSE)+
geom_errorbarh(data=d3,aes(y=jnd,xmin=meanJ-ciJ,xmax=meanJ+ciJ,color=coln),height=0.05,size=1.5)+ #X軸方向のエラーバー
scale_color_manual(values=cols)+
guides(colour=FALSE) #凡例を消す
plot(g1)
ggsave("fitdemo5.png", g1)
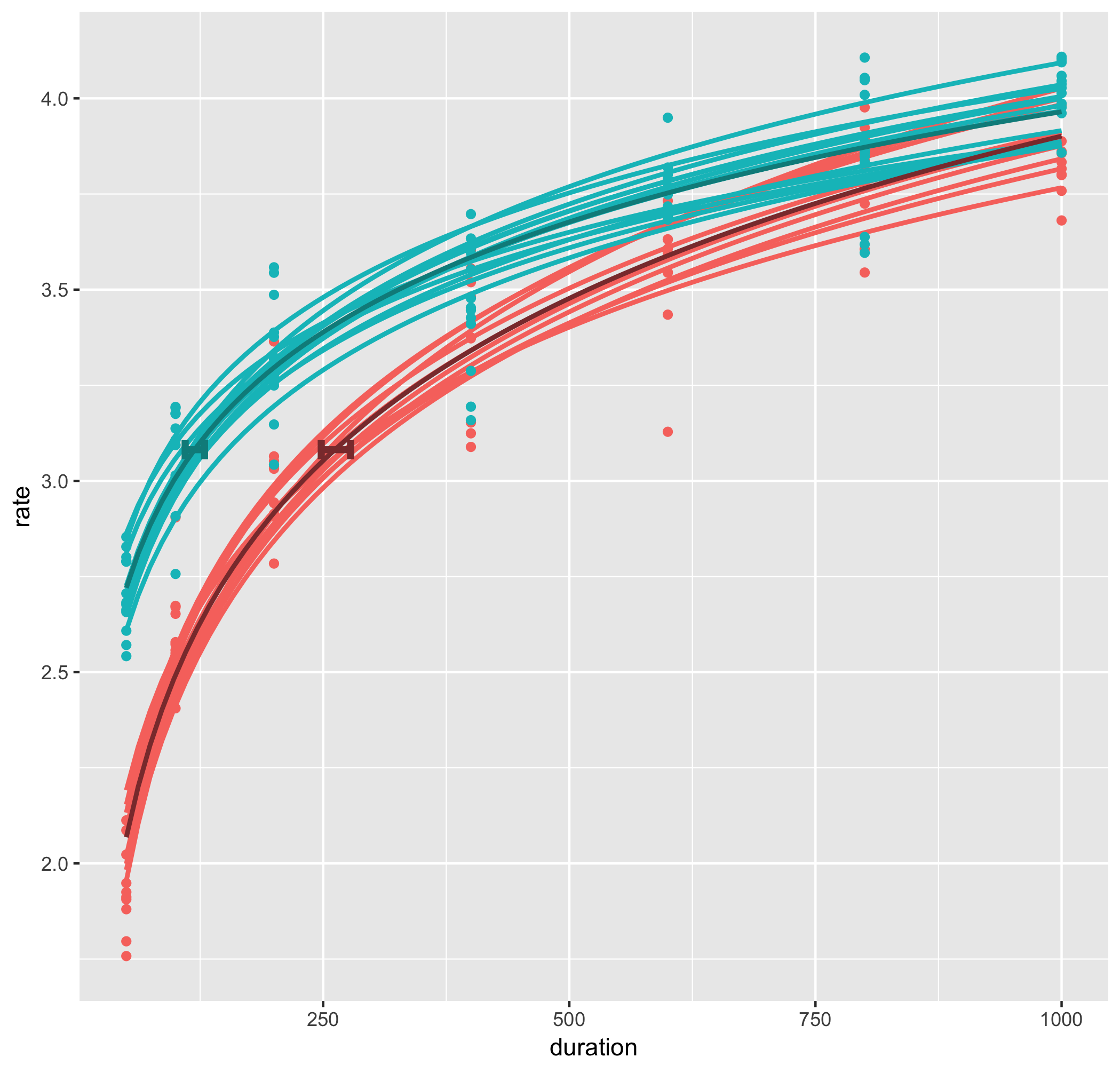
平均の近似曲線と水平方向のエラーバーの色を個人のプロットと分けました。
上の信頼区間の帯がついたプロットの例と比べると、個々人のラインプロットが透けていないせいで、エラーバーが見えづらいようです。
stat_smooth()では信頼区間の帯の透明度(alpha)しか変更できないので、geom_line()で線を描くことにします。
○ 各近似曲線の外観を調整する
各被験者のラインプロットについて、透明度を上げてみたいと思います。
個々人のJNDの算出とその平均値、信頼区間は算出しましたので、ggplotオブジェクトのみを再度作ります。
g2 <- ggplot()
for(i in 1 : length(subids)){
subd<-d[d$ID == subids[I], ]
g2 <- g2 + geom_line(data = subd, stat = "smooth",aes(duration, rate, color = group), method = "glm", formula = y ~ log(x), linetype = "solid", size = 2, alpha = .4)+
geom_point(data = subd, aes(duration, rate, color = group))
}
g2 <- g2 + geom_line(data = d2, stat = "smooth", aes(duration, rate, color = coln), method = "glm", formula = y ~ log(x), linetype = "solid", size = 2, alpha = .7)+
geom_errorbarh(data = d3, aes(y = jnd, xmin = meanJ - ciJ, xmax = meanJ + ciJ, color = coln), height = 0.05, size = 1.5)+
scale_color_manual(values = cols)+
guides(colour = FALSE)
plot(g2)
ggsave("fitdemo6.png", g2)
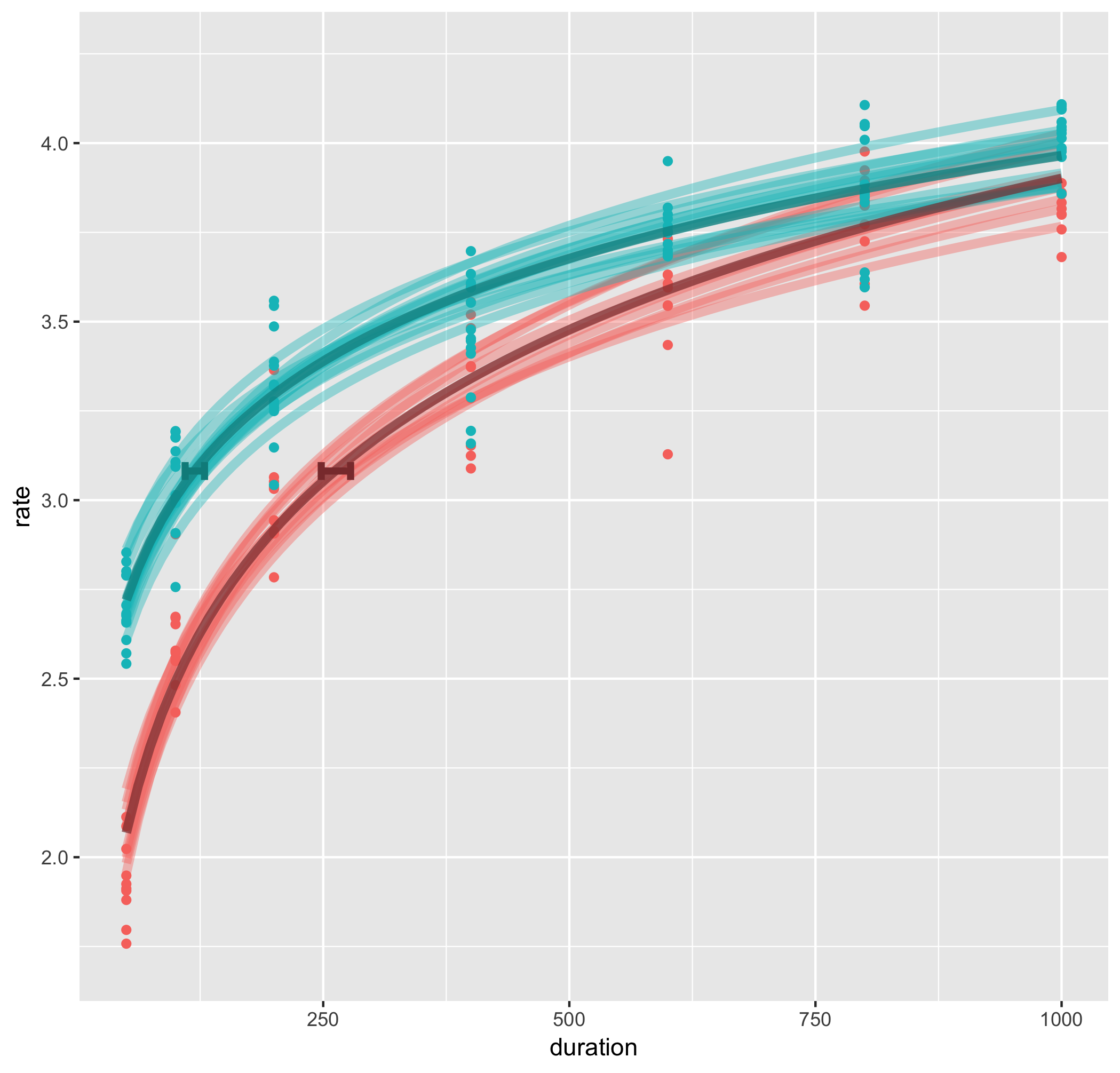
ちょっとはマシになったかな?